অ-সঙ্কেতী আর এন এ
এই নিবন্ধের ইংরেজি পরিভাষাগুলির বাংলা অনুবাদ আবশ্যক। নিবন্ধেরটির রচনা সংশোধনের প্রয়োজন হতে পারে। কারণ ব্যাকরণ, রচনাশৈলী, বানান বা বর্ণনাভঙ্গিগত সমস্যা রয়েছে। |
এই নিবন্ধটি অন্য একটি ভাষা থেকে আনাড়িভাবে অনুবাদ করা হয়েছে। এটি কোনও কম্পিউটার কর্তৃক অথবা দ্বিভাষিক দক্ষতাহীন কোনো অনুবাদক কর্তৃক অনূদিত হয়ে থাকতে পারে। |

একটি নন-কোডিং আরএনএ ( এনসিআরএনএ ) একটি আরএনএ অণু যা প্রোটিনে অনুবাদ হয় না। যে ডিএনএ ক্রম থেকে ক্রিয়ামূলক নন-কোডিং আরএনএ প্রতিলিপি হয় তাকে প্রায়শই আরএনএ জিন বলা হয় । অধিক প্রাপ্ত এবং গুরুত্বপূর্ণ নন-কোডিং RNAs গুলি হলো স্থানান্তর RNAs (tRNAs), রাইবোজোমাল RNAs (rRNAs), ছোটো RNA যেমন মাইক্রো RNA, siRNAs, piRNAs, snoRNAs, snRNAs, exRNAs, scaRNAs এবং দীর্ঘ ncRNAs (যেমন Xist এবং হটএয়ার ।)
মানব জিনোমের মধ্যে অ-সঙ্কেতী আরএনএর সংখ্যা অজানা; তবে সাম্প্রতিক ট্রান্সক্রিপ্টোমিক এবং বায়োইনফর্ম্যাটিক স্টাডিতে বোঝা গেছে যে তাদের হাজার হাজার রয়েছে। [১][২][৩][৪][৫] সদ্য চিহ্নিত অনেকগুলি এনসিআরএনএ কাজ শনাক্ত হয়নি [৬] এটিও সম্ভব হয় যে কিছু এনসিআরএনএ হয়তো অ-কার্যকরী (কখনও কখনও জাঙ্ক আরএনএ হিসাবে পরিচিত), এবং এটি কখনও কখনো ত্রুটিপূর্ণ (sprurious) ট্রান্সক্রিপশন এর পণ্য। [৭][৮]
নন-কোডিং আরএনএগুলি ক্যান্সার এবং আলঝাইমার সহ রোগগুলিতে অবদান রাখে বলে মনে করা হয়।
ইতিহাস এবং আবিষ্কার[সম্পাদনা]
নিউক্লিক অ্যাসিডগুলি প্রথম 1868 সালে ফ্রিডরিচ মাইশার [৯] দ্বারা আবিষ্কার করেছিলেন এবং ১৯৩৯ সালে আরএনএ প্রোটিন সংশ্লেষণে জড়িত ছিল বলে অনুমান করা হয়েছিলো। [১০] দুই দশক পরে, ফ্রান্সিস ক্রিক একটি কার্যকরী আরএনএ উপাদান ভবিষ্যদ্বাণী করেছিলেন যা অনুবাদকে মধ্যস্থতা করেছিল; তিনি যুক্তি দিয়েছিলেন যে এমআরএনএ ট্রান্সক্রিপ্টের সাথে বেস-জুটির পক্ষে একটি একটি খাঁটি পলিপেপটাইড এর চেয়ে একটি আরএনএ আরও উপযুক্ত। [১১]
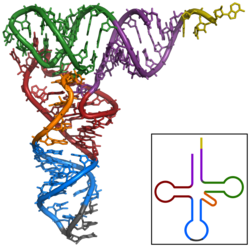
প্রথম শনাক্ত নন-কোডিং আরএনএটি ছিল বেকারের খামীরে পাওয়া একটি অ্যালানাইন টিআরএনএ, এর কাঠামোটি 1965 সালে প্রকাশিত হয়েছিল। [১২] শুদ্ধ অ্যালানাইন টিআরএনএ নমুনা তৈরি করতে রবার্ট ডব্লু। হোলি ও সহকর্মীবৃন্দ। বিশ্লেষণের জন্য মাত্র 1 গ্রাম পরিশোধিত টিআরএনএঅ্যালা দিতে ১৪০ কেজি বাণিজ্যিক বেকারের খামির ব্যবহার করেছেন। [১৩] ৮০ নিউক্লিওটাইড টি আর এন এ প্রথমে সিকোয়েন্স করা হয়েছিলো, প্রথমে অগ্ন্যাশয় ribonuclease দিয়ে পাচিত করে ( যা সাইটোসিন বা Uridine দিয়ে শেষ হওয়া টুকরা উৎপাদন করে) এবং তারপর takadiastase রাইবোনিউক্লিয়েজ TL ( যা Guanosine দিয়ে সমাপ্ত টুকরা উতপাদন করে)। ক্রোমাটোগ্রাফি এবং 5 এবং 3 'এর শেষগুলি সনাক্তকরণ এর পরে টুকরোগুলি আরএনএ ক্রমটি প্রতিষ্ঠিত করতে সহায়তা করেছিল। মূলত এই টিআরএনএর জন্য প্রস্তাবিত তিনটি কাঠামোর মধ্যে 'ক্লোভারলিফ' কাঠামোটি নিম্নলিখিত কয়েকটি প্রকাশনাতে স্বতন্ত্রভাবে প্রস্তাবিত হয়েছিল। [১৪][১৫][১৬][১৭] ১৯4৪ সালে দুটি স্বতন্ত্র গবেষণা গ্রুপ দ্বারা সম্পাদিত এক্স-রে ক্রিস্টালোগ্রাফিক বিশ্লেষণের পরে ক্লোভারলিফ গৌণ কাঠামোটি চূড়ান্ত করা হয়েছিল [১৮][১৯]
এর পরেই রাইবোসোমাল আরএনএ আবিষ্কার হয়েছিল , এর পরে 1980 এর দশকের প্রথম দিকে ইউআরএনএ সম্পরকে জানা গেছিল। তার পর থেকে, নতুন নন-কোডিং আরএনএগুলির আবিষ্কার স্নোআরএনএ, ক্সিস্ট, সিআরআইএসপিআর এবং আরও অনেকগুলি দিয়ে অব্যাহত রয়েছে। [২০] সাম্প্রতিক উল্লেখযোগ্য সংযোজনগুলির মধ্যে রয়েছে রাইবোসউইচস এবং এমআইআরএনএ ; পরবর্তীকালের সাথে সম্পর্কিত আরএনএআই পদ্ধতির আবিষ্কার ক্রেগ সি মেলো এবং অ্যান্ড্রু ফায়ারকে ফিজিওলজি বা মেডিসিনে 2006 সালের নোবেল পুরস্কার দিয়েছিল । [২১]
এনসিআরএনএর সাম্প্রতিক আবিষ্কারগুলি পরীক্ষামূলক এবং বায়োইনফরম্যাটিক উভয় পদ্ধতির মাধ্যমেই অর্জন করা হয়েছে।
জৈবিক ভূমিকা[সম্পাদনা]
ননকোডিং আরএনএ বিভিন্ন গোষ্ঠীর অন্তর্ভুক্ত এবং অনেক কোষীয় প্রক্রিয়াতে জড়িত। এগুলি অতি গুরুত্বপূর্ণ এনসিআরএনএ থেকে শুরু করে (যা বহু প্রজাতিতে একই রকম) ক্ষণস্থায়ী এনসিআরএনএর (যা কেবল কয়েকটি নিকটবর্তী প্রজাতিতে একই রকম)। আরও সংরক্ষিত এনসিআরএনএগুলি সর্বজনীন সাধারণ পূর্বপুরুষ এবং আরএনএ জগতের আণবিক জীবাশ্ম বা ধ্বংসাবশেষ বলে মনে করা হয় এবং তাদের বর্তমান ভূমিকা বেশিরভাগ ডিএনএ থেকে প্রোটিনের তথ্য প্রবাহ নিয়ন্ত্রণে থাকে। [২২][২৩][২৪]
অনুবাদনে[সম্পাদনা]

অনেকগুলি সংরক্ষিত, প্রয়োজনীয় প্রাচুর্যপূর্ণ এনসিআরএনএ অনুবাদে জড়িত। ribosomes নামক Ribonucleoprotein (RNP) গুলি হল কোষের প্রোটিন উৎপাদনের 'কারখানা' । রাইবোসোমে 60% এর বেশি রাইবোসোমাল আরএনএ থাকে ; এগুলি প্রোকারিওটিসে 3 টি এনসিআরএনএ এবং ইউক্যারিওটসে 4 টি এনসিআরএনএ দিয়ে গঠিত হয়। রাইবোসোমাল আরএনএগুলি অনুবাদনে অনুঘটকের কাজ করে। এনসিআরএনএর আরেকটি সেট, স্থানান্তর করণ আরএনএ, এমআরএনএ এবং প্রোটিনের মধ্যে একটি 'অ্যাডাপ্টার অণু' গঠন করে। এইচ / এসিএ বক্স এবং সি / ডি বক্স স্নোআরএনএ হ'ল এনসিআরএনএ যা আর্কিয়া এবং ইউকারিয়োটেসে পাওয়া যায়। আরএনেস এমআরপি ইউকারিয়োটসে সীমাবদ্ধ। এনসিআরএনএর উভয় গ্রুপই আরআরএনএর পরিপক্বতায় জড়িত। এস এন ও আর এন এ গুলি, এসএনআরএনএ আরআরএনএ, টিআরএনএ এর সমযোজী পরিবর্তনের দিকনির্দেশনা দেয় ; আরএনএজ এমআরপি, 18 এস এবং 5.8 এস আরআরএনএর মধ্যে অভ্যন্তরীণ স্পেসার 1 কে কেটে দেয়। সর্বব্যাপী এনসিআরএনএ, আর এন এজ পি, আরএনএজ এমআরপি-র একটি বিবর্তনীয় আত্মীয়। [২৬] আরএনএস পি পূর্ববর্তী-টিআরএনএ-এর 5'-পূর্ববর্তী (লিডার) উপাদানগুলি ক্লিভ করার মাধ্যমে টিআরএনএগুলির পরিপক্ব 5'-প্রান্ত তৈরি করে। SRP নামক আরেকটি সর্বব্যাপী RNP; জায়মান প্রোটিন কে চিনতে পারে এবং এন্ডোপ্লাজমিক রেটিকুলাম মধ্যে (ইউক্যারিয়োটস দের ক্ষেত্রে) এবং কোষ ঝিল্লি র মধ্যে (প্রোক্যারিওট দের ক্ষেত্রে) পরিবহন করে। ব্যাকটেরিয়া র ক্ষেত্রে পরিবাহী- বার্তাবাহী RNA- (tmRNA) একটি RNP যা , স্থগিত রাইবোজোম উদ্ধারকরণ, অসম্পূর্ণ পলিপেপটাইড ট্যাগিং এবং ত্রুটিপূর্ণ mRNA ক্ষয় ত্বরান্বিত করার কাজে জড়িত। [তথ্যসূত্র প্রয়োজন]
আরএনএ বিভক্তকরণে[সম্পাদনা]
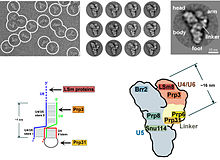
ইউক্যারিয়োটস দের ক্ষেত্রে spliceosome সঞ্চালিত করে স্প্লাইসিং প্রক্রিয়া, যা intron সিকোয়েন্স সরানোর জন্য অপরিহার্য, এই প্রক্রিয়া পরিপক্ব mRNA গঠনের জন্য প্রয়োজন বোধ করা হয়। স্প্লাইসোসোম হ'ল একটি আরএনপি যা প্রায়শই স্নারএনএনপি বা ত্রি-সানআরএনপি নামেও পরিচিত। স্প্লাইসোসোমের দুটি পৃথক রূপ রয়েছে, প্রধান এবং গৌণ রূপ। প্রধান স্প্লাইসোসোমের এনসিআরএনএ উপাদানগুলি হ'ল ইউ 1, ইউ 2, ইউ 4, ইউ 5 এবং ইউ 6 । ছোটোখাটো spliceosome এর ncRNA উপাদান ইউ 11, ইউ 12, ইউ 5, ইউ 4atac এবং ইউ 6atac । [তথ্যসূত্র প্রয়োজন]
আর একটি গোষ্ঠীর ইনট্রন, পোষক (সম্পূর্ণ) প্রতিলিপি থেকে তাদের নিজস্ব প্রতিলিপিগুলি অপসারণকে অনুঘটক করতে পারে; এগুলিকে স্ব-স্প্লিজিং আরএনএ বলা হয়। স্ব-বিভক্ত আরএনএ দুটি প্রধান গ্রুপ রয়েছে: গ্রুপ প্রথম অনুঘটক ইন্ট্রন এবং দ্বিতীয় গ্রুপ অনুঘটক ইন্ট্রন । এই এনসিআরএনএগুলি বহুধরনের জীবের এমআরএনএ, টিআরএনএ এবং আরআরএনএ পূর্ববর্তী অবস্থা থেকে তাদের নিজস্ব বিভাজনকে অনুঘটন করে। [তথ্যসূত্র প্রয়োজন]
স্তন্যপায়ী প্রাণীদের মধ্যে এটি দেখা গেছে যে স্নোআরএনএগুলি এমআরএনএর বিকল্প splicing নিয়ন্ত্রণ করতে পারে, উদাহরণস্বরূপ snoRNA HBII-52 সেরোটোনিন রিসেপ্টর 2 সি এর স্প্লাইসিং নিয়ন্ত্রণ করে। [২৭]
নেমাটোডগুলিতে, স্মাই এনসিআরএনএ এমআরএনএ ট্রান্স- স্প্লাইসিংয়ের সাথে জড়িত বলে মনে হয়। [তথ্যসূত্র প্রয়োজন]
ডিএনএর প্রতিরূপে[সম্পাদনা]

ওয়াই আরএনএ হ'ল স্টেম লুপ, যা ডিএনএর প্রতিরূপের জন্য প্রয়োজনীয়। উৎস স্বীকৃতি জটিল সহ ক্রোমাটিন এবং সূচনা প্রোটিনের সাথে আন্তঃক্রিয়ার মাধ্যমে এটি ডি এন এ প্রতিরূপে সহায়তা করে । [২৯][৩০] এগুলি Ro60 রাইবোনিউক্লিওপ্রোটিন কণার ও উপাদান [৩১] যা সিস্টেমিক লুপাস এরিথেমাটোসাস রোগীদের রোগীদের অটোইমিউন অ্যান্টিবডিগুলির লক্ষ্য। [৩২]
জিন নিয়ন্ত্রণে[সম্পাদনা]
অনেক হাজার জিনের অভিব্যক্তি এনসিআরএনএ দ্বারা নিয়ন্ত্রিত হয়। এই নিয়ন্ত্রণ ট্রান্স বা সিস হতে পারে। জানা গেছে যে একটি বিশেষ ধরনের এনসিআরএনএ, যাকে বলা হয় এনহ্যান্সার আরএনএ , জিনের বর্ধক অঞ্চল থেকে প্রতিলিপি হয়ে জিনের অভিব্যক্তি বিবর্ধনে কাজ করে। [তথ্যসূত্র প্রয়োজন]
ট্রান্স-আন্তঃক্রিয়া[সম্পাদনা]
উচ্চতর ইউক্যারিওটসে মাইক্রোআরএনএ জিনের এক্সপ্রেশন নিয়ন্ত্রণ করে। একটি একক এম আই আরএনএ শত শত জিনের প্রকাশের মাত্রা হ্রাস করতে পারে। সাধারণত বার্তাবাহী আর এন এ অণুর উপর আংশিক পরিপূরকতা র মাধ্যমে পরিণত এম আই আর এন এ কাজ করে (সাধারণতঃ 3 'ইউটিআরগুলিতে )। এম আই আর এন এ র প্রধান কাজ হল জিন অভিব্যক্তির মাত্রা কে কমানো।
এনসিআরএনএ আরএনএজ পিও জিনের এক্সপ্রেশনকে প্রভাবিত করে দেখা গেছে। মানব নিউক্লিয়াসে আরএনএস পি আরএনএ পলিমারেজ III দ্বারা লিখিত বিভিন্ন এনসিআরএনএর স্বাভাবিক এবং দক্ষ প্রতিলিপির জন্য প্রয়োজনীয়। এর মধ্যে রয়েছে টিআরএনএ, ৫ এস আরআরএনএ, এসআরপি আরএনএ এবং ইউ 6 এসএনআরএনএ জিন। আরএনএস পি, সক্রিয় টিআরএনএ জিন এবং 5 এস আরআরএনএ জিনের ক্রোমাটিনের সাথে পলিমারেজ-৩ সংযুক্তির মাধ্যমে ট্রানসক্রিপশনএ তার ভূমিকা পালন করে। [৩৩]
এটি দেখানো হয়েছে যে 7 এসকে আরএনএ, একটি মেটাজোয়ান এনসিআরএনএ, আরএনএ পলিমারেজ II ও প্রসারণ ফ্যাক্টর পি-টিইএফবির নেতিবাচক নিয়ামক হিসাবে কাজ করে এবং এই ক্রিয়াকলাপটি স্ট্রেস প্রতিক্রিয়া পথে অনুসরণ করে। [তথ্যসূত্র প্রয়োজন]
ব্যাকটিরিয়াল এনসিআরএনএ, 6 এস আরএনএ, নির্দিষ্টভাবে সিগমা 70 স্পেসিফিসিটি ফ্যাক্টর যুক্ত আরএনএ পলিমেরেজ হোলোএনজাইমের সাথে যুক্ত হয়। । [তথ্যসূত্র প্রয়োজন] এই ঘটনা, সিগমা- ৭০ -নির্ভর প্রমোটার কে বাধা দেয়।
আর একটি ব্যাকটিরিয় এনসিআরএনএ, অক্সি এস আরএনএ শাইন-ডালগার্নো সিকোয়েন্সগুলিকে আবদ্ধ করে রাইবোসোম বাইন্ডিং দ্বারা অনুবাদকে দমন করে। এসেরিচিয়া কোলিতে জারণ চাপের প্রতিক্রিয়ায় অক্সিজ আরএনএ প্ররোচিত হয়। [তথ্যসূত্র প্রয়োজন]
বি 2 আরএনএ হ'ল একটি ছোট, ননকডিং আরএনএ পলিমেরেজ-৩ প্রতিলিপি, যা ইঁদুরের কোষগুলিতে তাপের শকের প্রতিক্রিয়া হিসাবে এমআরএনএ ট্রান্সক্রিপশন দমন করে। বি 2 আরএনএ মূল পল-২ এর সাথে আবদ্ধ হয়ে প্রতিলিপি প্রতিরোধ করে। এই মিথস্ক্রিয়াটির মাধ্যমে, বি 2 আরএনএ প্রমোটারে প্রি-ইনিশিয়েশন কমপ্লেক্সগুলিতে মিলিত হয় এবং আরএনএ সংশ্লেষণকে অবরুদ্ধ করে। [৩৪]
সাম্প্রতিক একটি গবেষণায় দেখা গেছে যে কেবল এনসিআরএনএ সিকোয়েন্সের প্রতিলিখনের কাজটি জিনের অভিব্যক্তিতে প্রভাব ফেলতে পারে। স্কিজোস্যাকারোমাইসেস পম্বে ক্রোমাটিন পুনরাকৃতির জন্য এনসিআরএনএর আরএনএ পলিমেরেজ ২ প্রতিলিপি প্রয়োজন। বেশ কয়েকটি প্রজাতির এনসিআরএনএ যত প্রতিলিপি করা হয় তত ক্রোমাটিন ক্রমান্বয়ে একটি মুক্ত কনফিগারেশনে রূপান্তরিত হয় [৩৫]
সিস-আন্তঃক্রিয়া[সম্পাদনা]
অনেকগুলি এনসিআরএনএ প্রোটিন কোডিং জিনের 5 ' ইউটিআর (অ-অনূদিত অঞ্চলগুলি)তে অন্তর্নিহিত থাকে এবং বিভিন্নভাবে তাদের অভিব্যক্তিকে প্রভাবিত করে। উদাহরণস্বরূপ, একটি রাইবোসুইচ সরাসরি একটি ছোট লক্ষ্য অণুকে আবদ্ধ করতে পারে; লক্ষ্যটির সংযুক্তি জিনের ক্রিয়াকলাপকে প্রভাবিত করে। [তথ্যসূত্র প্রয়োজন]
আরএনএ লিডার সিকোয়েন্সগুলি অ্যামিনো অ্যাসিড বায়োসিন্থেটিক অপেরনের প্রথম জিনের পূর্বে পাওয়া যায়। এই আরএনএ উপাদানগুলি অপেরনের শেষ প্রান্তে অ্যামিনো অ্যাসিড সমৃদ্ধ খুব সংক্ষিপ্ত পেপটাইড সিকোয়েন্সগুলি এনকোডিং করা অঞ্চলে দুটি সম্ভাব্য কাঠামোর মধ্যে একটি গঠন করে। যখন নিয়ামক অ্যামিনো অ্যাসিডের আধিক্য থাকে তখন একটি টার্মিনেটর কাঠামো তৈরি হয় এবং লিডার ট্রান্সক্রিপ্টের উপর রাইবোসোম অগ্রগতি বাধাগ্রস্ত হয় না। যখন নিয়ামক অ্যামিনো অ্যাসিডের চার্জযুক্ত টিআরএনএর ঘাটতি থাকে তখন লিডার পেপটাইড থেমে যায় এবং অ্যান্টিটারমিনেটর কাঠামো গঠিত হয়। এটি আরএনএ পলিমেরেজকে অপেরন প্রতিলিপি করতে সহায়তা করে। পরিচিত আরএনএ লিডার রা হল হিস্টিডাইন অপেরন লিডার, লিউসিন অপেরন লিডার, থ্রেওনাইন অপেরন লিডার এবং ট্রিপটোফান অপেরন লিডার । [তথ্যসূত্র প্রয়োজন]
আয়রন রেসপন্স এলিমেন্টস (আইআরই) আয়রন রেসপন্স প্রোটিন (আইআরপি) দ্বারা আবদ্ধ হয়। আইআরই বিভিন্ন এমআরএনএর ইউটিআরগুলিতে পাওয়া যায় যার পণ্যগুলি লোহা বিপাকের সাথে জড়িত। যখন আয়রনের ঘনত্ব কম থাকে, আইআরপিগুলি ফেরিটিন এমআরএনএ আইআরই-কে বেঁধে দিয়ে অনুবাদ দমন করে। [তথ্যসূত্র প্রয়োজন]
অভ্যন্তরীণ রাইবোসোম এন্ট্রি সাইটগুলি (আইআরইএস) হ'ল আরএনএ স্ট্রাকচার যা একটি একটি এম আর এন এ অণুর মধ্যিখান থেকে ট্রান্সলেশনে সহায়তা করে। [তথ্যসূত্র প্রয়োজন]
জিনোম প্রতিরক্ষায়[সম্পাদনা]
পিউই-আন্তঃকারী RNAs (piRNAs) স্তন্যপায়ী র টেস্টিস এবং দেহকোষে প্রকাশ হয় এবং Piwi প্রোটিনসমূহর সাথে RNA-প্রোটিন কমপ্লেক্স গঠন করে। এই পাই আরএনএ কমপ্লেক্সগুলি (পিআইআরসি) জনন রেখার কোষগুলিতে রেট্রোট্রান্সপসসন এবং অন্যান্য জিনগত উপাদানগুলির ট্রান্সক্রিপশনাল জিনকে সাইলেন্টিংয়ের সাথে যুক্ত বলে মনে করা হয়েছে, বিশেষত স্পারমাটোজেনেসিস প্রক্রিয়ায় ।
ক্লাস্টার্ড নিয়মিতভাবে ইন্টারস্টেপড শর্ট প্যালিনড্রমিক রিপিটস (সিআরআইএসপিআর) অনেকগুলি ব্যাকটিরিয়া এবং আর্চিয়ার ডিএনএতে পুনরাবৃত্তি হয়। পুনরাবৃত্তিগুলি একই দৈর্ঘ্যের স্পেসার দ্বারা পৃথক করা হয়। এটি প্রদর্শিত হয়েছে যে এই স্পেসারগুলি ফাজ ভাইরাস থেকে নেওয়া যেতে পারে এবং পরবর্তীকালে কোষকে সংক্রমণ থেকে রক্ষা করতে সহায়তা করে।
ক্রোমোজোম স্ট্রাকচার[সম্পাদনা]
টেলোমারেজ একটি RNP এনজাইম নির্দিষ্ট যোগ ডিএনএ অনুক্রম পুনরাবৃত্তি ( "TTAGGG" মেরুদন্ডী মধ্যে) এর টেলোমেরিক অঞ্চল, ইউক্যারিওটিক প্রান্ত পাওয়া যা ক্রোমোজমের । টেলোমিরেসে কনডেন্সড ডিএনএ উপাদান থাকে, ক্রোমোজোমগুলিকে স্থিতিশীলতা দেয়। এনজাইম হ'ল বিপরীত ট্রান্সক্রিপিটস যা টেলোমারেজ আরএনএ বহন করে, এটি টেম্পলেট হিসাবে ব্যবহৃত হয় যখন এটি টেলোমেয়ারগুলি দীর্ঘায়িত করে, যা প্রতিটি প্রতিলিপি চক্রের পরে সংক্ষিপ্ত করা হয় ।
এক্সিস্ট (এক্স- অ্যাক্টিভ -নির্দিষ্ট ট্রান্সক্রিপ্ট) প্ল্যাসেন্টাল স্তন্যপায়ী প্রাণীদের এক্স ক্রোমোজোমে একটি দীর্ঘ এনসিআরএনএ জিন যা বার ক্রমোসোম নিষ্ক্রিয়করণ প্রক্রিয়াটির প্রধান প্রভাবশালী হিসাবে কাজ করে বারের দেহ গঠন করে। একটি এন্টিসেন্স আরএনএ, টিসিক্স, এক্সের নেতিবাচক নিয়ামক। সিক্স এক্সপ্রেশন অভাবযুক্ত এক্স ক্রোমোসোমগুলি (এবং এইভাবে এক্সস্ট ট্রান্সক্রিপশন উচ্চ স্তরের রয়েছে) সাধারণ ক্রোমোসোমের চেয়ে বেশি ঘন ঘন নিষ্ক্রিয় হয়। ড্রোসোফিলিডগুলিতে, যা একটি এক্সওয়াই লিঙ্গ-নির্ধারণ ব্যবস্থাও ব্যবহার করে, ডক্স ক্ষতিপূরণে রক্স (এক্সের উপর আরএনএ) আরএনএ জড়িত। [৩৬] Xist এবং roX উভয়ই হিস্টোন-সংশোধনকারী এনজাইমদের নিয়োগের মাধ্যমে প্রতিলিপিটির এপিজেনেটিক নিয়ন্ত্রণের মাধ্যমে পরিচালনা করে।
দ্বৈত-ফাংশন আরএনএ[সম্পাদনা]
বাই ফাংশনাল আরএনএ বা দ্বৈত-ফাংশন আরএনএ হ'ল দুটি পৃথক ফাংশনযুক্ত আরএনএ । [৩৭][৩৮] পরিচিত দ্বিখণ্ডিত আরএনএর বেশিরভাগ হ'ল এমআরএনএ যা প্রোটিন এবং এনসিআরএনএ উভয়কেই এনকোড করে। তবে, ক্রমবর্ধমান এনসিআরএনএ দুটি পৃথক এনসিআরএনএ বিভাগে পড়ে; যেমন, এইচ / এসিএ বক্স স্নোআরএনএ এবং এমআইআরএনএ । [৩৯][৪০]
দ্বিখণ্ডিত আরএনএর দুটি সুপরিচিত উদাহরণ হ'ল এসজিআরএস আরএনএ এবং আরএনএ-৩ । তবে, মুষ্টিমেয় অন্যান্য দ্বিখণ্ডিত আরএনএগুলি (যেমন, স্টেরয়েড রিসেপ্টর অ্যাক্টিভেটর / এসআরএ,[৪১] VegT আরএনএ,[৪২][৪৩][৪৪] ওসকার আরএনএ,[৪৪] ENOD40,[৪৫] পি53 আরএনএ [৪৬] এবং এসআর 1 আরএনএ । [৪৭] । [৪৮]
হরমোন হিসাবে[সম্পাদনা]
নির্দিষ্ট নন-কোডিং আরএনএ এবং হরমোন-নিয়ন্ত্রিত পথগুলির নিয়ন্ত্রণের মধ্যে একটি গুরুত্বপূর্ণ লিঙ্ক রয়েছে। ড্রসোফিলায়, একডিসোন এবং জুভেনাইল হরমোন জাতীয় হরমোনগুলি নির্দিষ্ট এমআরএনএ- এর প্রকাশকে উৎসাহিত করতে পারে। তদ্ব্যতীত, এই নিয়ম সি এর মধ্যে স্বতন্ত্র টেম্পোরাল পয়েন্টগুলিতে ঘটে থাকে এলিগানসের বিকাশ। [৪৯] স্তন্যপায়ী তে মীর-206 এর একটি অত্যন্ত গুরুত্বপূর্ণ ইস্ট্রজেন -receptor-আলফা র নিয়ন্ত্রক। [৫০]
নন-কোডিং আরএনএগুলি বেশ কয়েকটি অন্তঃস্রাবের অঙ্গগুলির বিকাশের পাশাপাশি ডায়াবেটিস মেলিটাসের মতো অন্তঃস্রাবজনিত রোগে গুরুত্বপূর্ণ। [৫১] বিশেষ করে এমসিএফ-7 সেল লাইনে, 17β- যোগে estradiol এসব নন-কোডিং RNAs ইস্ট্রজেন-সক্রিয় কোডিং জিন কাছাকাছি lncRNAs নামক বিশ্বব্যাপী ট্রান্সক্রিপশন বেড়েছে। [৫২]
রোগের ভূমিকা[সম্পাদনা]
প্রোটিনের মতো, দেহের অভ্যন্তরে এনসিআরএনএ এর পুস্তকে প্রোটিন বা ভারসাম্যহীনতা বিভিন্ন ধরনের রোগের কারণ হতে পারে।
কর্কট[সম্পাদনা]
অনেক এনসিআরএনএ ক্যান্সারযুক্ত টিস্যুগুলিতে অস্বাভাবিক প্রকাশের ধরনগুলি দেখায়। [৪] এর মধ্যে এমআইআরএনএ, লম্বা এমআরএনএ-জাতীয় এনসিআরএনএ,[৫৩][৫৪] জিএএস৫ [৫৫][৫৫] এসএনআরআর্ড 50,[৫৬] টেলোমারেজ আরএনএ এবং ওয়াই আরএনএ অন্তর্ভুক্ত রয়েছে । [৫৭] মাইআরএনএগুলি অনেকগুলি প্রোটিন কোডিং জিনের বৃহত আকারের নিয়ন্ত্রণে জড়িত,[৫৮][৫৯] ডিএনএ প্রতিরূপের সূচনার জন্য ওয়াই আরএনএগুলি গুরুত্বপূর্ণ,[২৯] টেলোমেরাজ আরএনএ, যা টেলোমারেজের জন্য প্রাইমারের কাজ করে, একটি আরএনপি যা প্রসারিত ক্রোমোসোম প্রান্তে টেলোমে্রিক অঞ্চলগুলি (আরও তথ্যের জন্য টেলোমেরেস এবং রোগ দেখুন)। দীর্ঘ এমআরএনএ-জাতীয় এনসিআরএনএর সরাসরি কাজটি কম স্পষ্ট।
মাইআর -১-1-১ এবং এমআইআর -15 প্রাথমিক পূর্ববর্তীগুলিতে জননকোষ-রেখা পরিবর্তনগুলি নিয়ন্ত্রণের জনসংখ্যার তুলনায় দীর্ঘস্থায়ী লিম্ফোসাইটিক লিউকেমিয়ায় আক্রান্ত রোগীদের মধ্যে অনেক বেশি ঘন ঘন দেখা গেছে। [৬০][৬১]
এটির পরামর্শ দেওয়া হয়েছে যে একটি বিরল এসএনপি ( আরএস 11614913 ) যে মির -196a2- এর ওভারল্যাপ রয়েছে সেগুলি অ-ছোট কোষ ফুসফুস কার্সিনোমার সাথে জড়িত বলে প্রমাণিত হয়েছে। [৬২] তেমনি, 17 টি মাইআরএনএ-র একটি স্ক্রিন যা স্তন ক্যান্সার সম্পর্কিত জিনগুলির বেশ কয়েকটি নিয়ন্ত্রণ করার পূর্বাভাস দিয়েছিল তারা মাইক্রোআরএনএ'র এমআইআর -17 এবং মাইআর -30 সি -1 এফ রোগীদের মধ্যে পার্থক্য খুঁজে পেয়েছে; এই রোগীদের বিআরসিএ 1 বা বিআরসিএ 2 রূপান্তরগুলির অবিবাহিত ছিল, এই এমআরএনএগুলির পরিবর্তনের কারণে পারিবারিক স্তনের ক্যান্সার হওয়ার সম্ভাবনা ধার দেয়। [৬৩] পি 53 হল টিউমার দমনকারী এবং টিউমার গঠন এবং অগ্রগতি প্রতিরোধ সবচেয়ে গুরুত্বপূর্ণ এজেন্ট। P53 প্রোটিন সেলুলার স্ট্রেস প্রতিক্রিয়া বাড়াতে একটি গুরুত্বপূর্ণ ভূমিকা সহ প্রতিলিপি ফ্যাক্টর হিসাবে কাজ করে। ক্যান্সারে তার গুরুত্বপূর্ণ ভূমিকা ছাড়াও, পি 53 ডায়াবেটিস, ইস্কেমিয়ার পরে কোষের মৃত্যু এবং হান্টিংটন, পার্কিনসন এবং আলৎসহাইমারের রোগ-এর মতো বিভিন্ন নিউরোডিজেনারেটিভ রোগ সহ অন্যান্য রোগে জড়িত রয়েছে। অধ্যয়নগুলিতে পরামর্শ দেওয়া হয়েছে যে পি 53 এক্সপ্রেশনটি কোডিং নন-কোডিং আরএনএ দ্বারা নিয়ন্ত্রণের সাপেক্ষে। [৩]
প্রডার – উইল সিন্ড্রোম[সম্পাদনা]
সি / ডি বাক্সের স্নোআরএনএ এসএনআরডি 116 এর 48 টি অনুলিপি মুছে ফেলা প্রাদার – উইল সিনড্রোমের প্রাথমিক কারণ হিসাবে দেখা গেছে। [৬৪][৬৫][৬৬][৬৭] প্রেডার – উইল একটি বিকাশযুক্ত ব্যাধি যা অতিরিক্ত খাওয়া এবং শেখার অসুবিধার সাথে জড়িত। SNORD116 এর বেশ কয়েকটি প্রোটিন-কোডিং জিনের মধ্যে সম্ভাব্য টার্গেট সাইট রয়েছে এবং বিকল্প স্প্লাইকিং নিয়ন্ত্রণে ভূমিকা রাখতে পারে। [৬৮]
অটিজম[সম্পাদনা]
ছোট নিউক্লোলার আরএনএ এসএনআরড 115 জিন ক্লাস্টারযুক্ত ক্রোমোসামাল লোকাসকে প্রায় 5% অটিস্টিক বৈশিষ্ট্যযুক্ত ব্যক্তিতে ডুপ্লিকেটেড অবস্থায় পাওয়া গেছে। [৬৯][৭০] SNORD115 ক্লাস্টারের নকল থাকতে ইঞ্জিনিয়ার করা একটি মাউস মডেল অটিস্টিক-জাতীয় আচরণ প্রদর্শন করে। [৭১] সাম্প্রতিক ময়না মস্তিষ্কের টিস্যুগুলির একটি ছোট্ট গবেষণায় নিয়ন্ত্রণের তুলনায় অটিস্টিক মস্তিষ্কের প্রিফ্রন্টাল কর্টেক্স এবং সেরিবেলিয়ামে দীর্ঘ নন-কোডিং আরএনএগুলির পরিবর্তিত অভিব্যক্তি প্রদর্শিত হয়েছিল। [৭২]
কার্টিজ – চুলের হাইপোপ্লাজিয়া[সম্পাদনা]
মধ্যে পরিব্যক্তি RNase এমআরপি কারণ দেখানো হয়েছে তরুণাস্থি-চুল hypoplasia, যেমন শর্ট মর্যাদা, বিক্ষিপ্ত চুল, কঙ্কাল অস্বাভাবিকতা এবং একটি চাপা ইমিউন সিস্টেম যে মধ্যে ঘন হিসাবে উপসর্গের একটি অ্যারের সঙ্গে যুক্ত একটি রোগ অ্যামিশ এবং ফিনিশ । [৭৩][৭৪][৭৫] সেরা ঘটায়, বৈকল্পিক একটি A টু জি হয় রূপান্তরটি নিউক্লিওটাইড 70 একটি লুপ অঞ্চলে যার এমন কোন দুই ক্ষার 5 ' সংরক্ষিত pseudoknot । তবে আরএনজে এমআরপি-র মধ্যে আরও অনেকগুলি মিউটেশনও সিএইচএইচ সৃষ্টি করে।
আলৎসহাইমারের রোগ[সম্পাদনা]
অ্যান্টিসেন্স আরএনএ, BACE1-AS বিপরীত স্ট্র্যান্ড থেকে BACE1 এ প্রতিলিপি করা হয় এবং আলঝাইমার রোগে আক্রান্ত রোগীদের মধ্যে এটি আপরেগুলেটেড হয়। [৭৬] BACE1-AS BACE1 এমআরএনএ স্থিতিশীলতা বৃদ্ধি করে এবং ট্রান্সক্রিপশনোত্তর পরবর্তী ফিড-ফরওয়ার্ড প্রক্রিয়াটির মাধ্যমে অতিরিক্ত BACE1 উৎপন্ন করে BACE1 এর অভিব্যক্তি নিয়ন্ত্রণ করে। একই প্রক্রিয়া দ্বারা এটি বোকা প্লেকগুলির মূল উপাদান বিটা অ্যামাইলয়েডের ঘনত্ব বাড়ায়। আলএইইমার রোগের বিষয়গুলিতে এবং অ্যামাইলয়েড পূর্ববর্তী প্রোটিন ট্রান্সজেনিক ইঁদুরগুলিতে BACE1-AS ঘনত্বকে উন্নত করা হয়।
miR-96 এবং শ্রবণশক্তি হ্রাস[সম্পাদনা]
পরিপক্ব এমআইআর -96 এর বীজ অঞ্চলের মধ্যে পার্থক্যটি অটোজোমাল ডোমিন্যান্ট , মানুষ এবং ইঁদুরের প্রগতিশীল শ্রবণ ক্ষতির সাথে যুক্ত। সমজাতীয় মিউট্যান্ট ইঁদুরগুলি গভীরভাবে বধির ছিল, কোনও কোক্লিয়ার প্রতিক্রিয়া দেখায় না। ভিন্ন ভিন্ন ইঁদুর এবং মানুষ ক্রমান্বয়ে শোনার ক্ষমতা হারাতে থাকে। [৭৭][৭৮][৭৯]
ক্রিয়ামূলক আরএনএ (এফআরএনএ) এবং এনসিআরএনএ মধ্যে পার্থক্য[সম্পাদনা]
আরএনএ স্তরে কার্যকরী অঞ্চলগুলি বর্ণনা করতে যেগুলি একা একা থাকা আরএনএ ট্রান্সক্রিপ্টগুলি হতে পারে বা নাও হতে পারে বিজ্ঞানীরা এনসিআরএনএ থেকে ফাংশনাল আরএনএ ( এফআরএনএ ) আলাদা করতে শুরু করেছেন। [৮০][৮১][৮২] এর দ্বারা বোঝা যায় যে এফআরএনএ (যেমন রাইবোসউইচস, সিসিসআইএস উপাদান এবং অন্যান্য সিস-রেগুলেটরি অঞ্চল) এনসিআরএনএ নয়। তা সত্ত্বেও fRNA এছাড়াও অন্তর্ভুক্ত হতে পারে mRNA, এই দেখেন, আরএনএ প্রোটিন জন্য কোডিং, এবং অত: পর কার্যকরী হয় না। অতিরিক্তভাবে কৃত্রিমভাবে বিকশিত আরএনএগুলিও এফআরএনএ ছাতার মেয়াদে পড়ে। কিছু প্রকাশনা [২০] বলেছে যে এনসিআরএনএ এবং এফআরএনএ প্রায় সমার্থক, তবে অন্যরা উল্লেখ করেছেন যে এনোটোটেড এনসিআরএনএর একটি বৃহত অংশের সম্ভবত কোনও কার্যকারিতা নেই। [৭][৮] কোয়ালিফায়ার এমআরএনএ দ্বারা প্রোটিন কোডিং আরএনএ ( ম্যাসেঞ্জার আরএনএ ) এর পার্থক্য ইতোমধ্যে দেওয়া হয়েছে বলে কেবল আরএনএ শব্দটি ব্যবহার করার পরামর্শ দেওয়া হয়েছে। [৮৩] এটি কোনও জিনকে "নন-কোডিং এনকোডিং" আরএনএ করার সময় অস্পষ্টতা দূর করে। এছাড়াও, প্রকাশিত সাহিত্য এবং ডেটাসেটগুলিতে অপ্রকাশিত বেশ কয়েকটি এনসিআরএনএ থাকতে পারে। [৮৪][৮৫][৮৬]
আরও দেখুন[সম্পাদনা]
- এক্সট্রা সেলুলার আরএনএ
- আরএনএগুলির তালিকা
- নিউক্লিক অ্যাসিড গঠন
- আরফাম (Rfam)
- রিবোসউইচ
- রিবোজাইম
- পরিবেশগত নমুনায় উপস্থিত আরএনএরা
- ভিএ (ভাইরাল সম্পর্কিত) আরএনএ
তথ্যসূত্র[সম্পাদনা]
- ↑ Cheng J, Kapranov P, Drenkow J, Dike S, Brubaker S, Patel S, Long J, Stern D, Tammana H, Helt G, Sementchenko V, Piccolboni A, Bekiranov S, Bailey DK, Ganesh M, Ghosh S, Bell I, Gerhard DS, Gingeras TR (মে ২০০৫)। "Transcriptional maps of 10 human chromosomes at 5-nucleotide resolution": 1149–54। ডিওআই:10.1126/science.1108625। পিএমআইডি 15790807।
- ↑ Washietl S, Pedersen JS, Korbel JO, Stocsits C, Gruber AR, Hackermüller J, Hertel J, Lindemeyer M, Reiche K, Tanzer A, Ucla C, Wyss C, Antonarakis SE, Denoeud F, Lagarde J, Drenkow J, Kapranov P, Gingeras TR, Guigó R, Snyder M, Gerstein MB, Reymond A, Hofacker IL, Stadler PF (জুন ২০০৭)। "Structured RNAs in the ENCODE selected regions of the human genome": 852–64। ডিওআই:10.1101/gr.5650707। পিএমআইডি 17568003। পিএমসি 1891344
 ।
।
- ↑ ক খ Morris KV, সম্পাদক (২০১২)। Non-coding RNAs and Epigenetic Regulation of Gene Expression: Drivers of Natural Selection। Caister Academic Press। আইএসবিএন 978-1-904455-94-3।
- ↑ ক খ Shahrouki P, Larsson E (২০১২)। "The non-coding oncogene: a case of missing DNA evidence?": 170। ডিওআই:10.3389/fgene.2012.00170। পিএমআইডি 22988449। পিএমসি 3439828
 ।
।
- ↑ van Bakel H, Nislow C, Blencowe BJ, Hughes TR (মে ২০১০)। "Most "dark matter" transcripts are associated with known genes": e1000371। ডিওআই:10.1371/journal.pbio.1000371। পিএমআইডি 20502517। পিএমসি 2872640
 ।
।
- ↑ Hüttenhofer A, Schattner P, Polacek N (মে ২০০৫)। "Non-coding RNAs: hope or hype?": 289–97। ডিওআই:10.1016/j.tig.2005.03.007। পিএমআইডি 15851066।
- ↑ ক খ Brosius J (মে ২০০৫)। "Waste not, want not--transcript excess in multicellular eukaryotes": 287–8। ডিওআই:10.1016/j.tig.2005.02.014। পিএমআইডি 15851065।
- ↑ ক খ Palazzo AF, Lee ES (২০১৫)। "Non-coding RNA: what is functional and what is junk?": 2। ডিওআই:10.3389/fgene.2015.00002। পিএমআইডি 25674102। পিএমসি 4306305
 ।
।
- ↑ Dahm R (ফেব্রুয়ারি ২০০৫)। "Friedrich Miescher and the discovery of DNA": 274–88। ডিওআই:10.1016/j.ydbio.2004.11.028। পিএমআইডি 15680349।
- ↑ Caspersson T, Schultz J (১৯৩৯)। "Pentose nucleotides in the cytoplasm of growing tissues": 602–3। ডিওআই:10.1038/143602c0।
- ↑ Crick FH (১৯৫৮)। "On protein synthesis": 138–63। পিএমআইডি 13580867।
- ↑ Holley RW, Apgar J, Everett GA, Madison JT, Marquisee M, Merrill SH, Penswick JR, Zamir A (মার্চ ১৯৬৫)। "Structure of a Ribonucleic Acid": 1462–5। ডিওআই:10.1126/science.147.3664.1462। পিএমআইডি 14263761।
- ↑ "The Nobel Prize in Physiology or Medicine 1968"। Nobel Foundation। সংগ্রহের তারিখ ২০০৭-০৭-২৮।
- ↑ Madison JT, Everett GA, Kung H (জুলাই ১৯৬৬)। "Nucleotide sequence of a yeast tyrosine transfer RNA": 531–4। ডিওআই:10.1126/science.153.3735.531। পিএমআইডি 5938777। সাইট সিয়ারX 10.1.1.1001.2662
 ।
।
- ↑ Zachau HG, Dütting D, Feldmann H, Melchers F, Karau W (১৯৬৬)। "Serine specific transfer ribonucleic acids. XIV. Comparison of nucleotide sequences and secondary structure models": 417–24। ডিওআই:10.1101/SQB.1966.031.01.054। পিএমআইডি 5237198।
- ↑ Dudock BS, Katz G, Taylor EK, Holley RW (মার্চ ১৯৬৯)। "Primary structure of wheat germ phenylalanine transfer RNA": 941–5। ডিওআই:10.1073/pnas.62.3.941। পিএমআইডি 5257014। পিএমসি 223689
 ।
।
- ↑ Cramer F, Doepner H, Haar F VD, Schlimme E, Seidel H (ডিসেম্বর ১৯৬৮)। "On the conformation of transfer RNA": 1384–91। ডিওআই:10.1073/pnas.61.4.1384। পিএমআইডি 4884685। পিএমসি 225267
 ।
।
- ↑ Ladner JE, Jack A, Robertus JD, Brown RS, Rhodes D, Clark BF, Klug A (নভেম্বর ১৯৭৫)। "Structure of yeast phenylalanine transfer RNA at 2.5 A resolution": 4414–8। ডিওআই:10.1073/pnas.72.11.4414। পিএমআইডি 1105583। পিএমসি 388732
 ।
।
- ↑ Kim SH, Quigley GJ, Suddath FL, McPherson A, Sneden D, Kim JJ, Weinzierl J, Rich A (জানুয়ারি ১৯৭৩)। "Three-dimensional structure of yeast phenylalanine transfer RNA: folding of the polynucleotide chain": 285–8। ডিওআই:10.1126/science.179.4070.285। পিএমআইডি 4566654।
- ↑ ক খ Eddy SR (ডিসেম্বর ২০০১)। "Non-coding RNA genes and the modern RNA world": 919–29। ডিওআই:10.1038/35103511। পিএমআইডি 11733745।
- ↑ Daneholt, Bertil। "Advanced Information: RNA interference"। The Nobel Prize in Physiology or Medicine 2006। ২০০৭-০১-২০ তারিখে মূল থেকে আর্কাইভ করা। সংগ্রহের তারিখ ২০০৭-০১-২৫।
- ↑ Jeffares DC, Poole AM, Penny D (জানুয়ারি ১৯৯৮)। "Relics from the RNA world": 18–36। ডিওআই:10.1007/PL00006280। পিএমআইডি 9419222।
- ↑ Poole AM, Jeffares DC, Penny D (জানুয়ারি ১৯৯৮)। "The path from the RNA world": 1–17। ডিওআই:10.1007/PL00006275। পিএমআইডি 9419221।
- ↑ Poole A, Jeffares D, Penny D (অক্টোবর ১৯৯৯)। "Early evolution: prokaryotes, the new kids on the block": 880–9। ডিওআই:10.1002/(SICI)1521-1878(199910)21:10<880::AID-BIES11>3.0.CO;2-P। পিএমআইডি 10497339।
- ↑ Ban N, Nissen P, Hansen J, Moore PB, Steitz TA (আগস্ট ২০০০)। "The complete atomic structure of the large ribosomal subunit at 2.4 A resolution": 905–20। ডিওআই:10.1126/science.289.5481.905। পিএমআইডি 10937989। সাইট সিয়ারX 10.1.1.58.2271
 ।
।
- ↑ Zhu Y, Stribinskis V, Ramos KS, Li Y (মে ২০০৬)। "Sequence analysis of RNase MRP RNA reveals its origination from eukaryotic RNase P RNA": 699–706। ডিওআই:10.1261/rna.2284906। পিএমআইডি 16540690। পিএমসি 1440897
 ।
।
- ↑ Kishore S, Stamm S (জানুয়ারি ২০০৬)। "The snoRNA HBII-52 regulates alternative splicing of the serotonin receptor 2C": 230–2। ডিওআই:10.1126/science.1118265। পিএমআইডি 16357227।
- ↑ Stein AJ, Fuchs G, Fu C, Wolin SL, Reinisch KM (মে ২০০৫)। "Structural insights into RNA quality control: the Ro autoantigen binds misfolded RNAs via its central cavity": 529–39। ডিওআই:10.1016/j.cell.2005.03.009। পিএমআইডি 15907467। পিএমসি 1769319
 ।
।
- ↑ ক খ Christov CP, Gardiner TJ, Szüts D, Krude T (সেপ্টেম্বর ২০০৬)। "Functional requirement of noncoding Y RNAs for human chromosomal DNA replication": 6993–7004। ডিওআই:10.1128/MCB.01060-06। পিএমআইডি 16943439। পিএমসি 1592862
 ।
।
- ↑ Zhang AT, Langley AR, Christov CP, Kheir E, Shafee T, Gardiner TJ, Krude T (জুন ২০১১)। "Dynamic interaction of Y RNAs with chromatin and initiation proteins during human DNA replication": 2058–69। ডিওআই:10.1242/jcs.086561। পিএমআইডি 21610089। পিএমসি 3104036
 ।
।
- ↑ Hall AE, Turnbull C, Dalmay T (এপ্রিল ২০১৩)। "Y RNAs: recent developments": 103–10। ডিওআই:10.1515/bmc-2012-0050। পিএমআইডি 25436569।
- ↑ Lerner MR, Boyle JA, Hardin JA, Steitz JA (জানুয়ারি ১৯৮১)। "Two novel classes of small ribonucleoproteins detected by antibodies associated with lupus erythematosus": 400–2। ডিওআই:10.1126/science.6164096। পিএমআইডি 6164096।
- ↑ Reiner R, Ben-Asouli Y, Krilovetzky I, Jarrous N (জুন ২০০৬)। "A role for the catalytic ribonucleoprotein RNase P in RNA polymerase III transcription": 1621–35। ডিওআই:10.1101/gad.386706। পিএমআইডি 16778078। পিএমসি 1482482
 ।
।
- ↑ Espinoza CA, Allen TA, Hieb AR, Kugel JF, Goodrich JA (সেপ্টেম্বর ২০০৪)। "B2 RNA binds directly to RNA polymerase II to repress transcript synthesis": 822–9। ডিওআই:10.1038/nsmb812। পিএমআইডি 15300239।
- ↑ Hirota K, Miyoshi T, Kugou K, Hoffman CS, Shibata T, Ohta K (নভেম্বর ২০০৮)। "Stepwise chromatin remodelling by a cascade of transcription initiation of non-coding RNAs": 130–4। ডিওআই:10.1038/nature07348। পিএমআইডি 18820678।
- ↑ Park Y, Kelley RL, Oh H, Kuroda MI, Meller VH (নভেম্বর ২০০২)। "Extent of chromatin spreading determined by roX RNA recruitment of MSL proteins": 1620–3। ডিওআই:10.1126/science.1076686। পিএমআইডি 12446910।
- ↑ Wadler CS, Vanderpool CK (ডিসেম্বর ২০০৭)। "A dual function for a bacterial small RNA: SgrS performs base pairing-dependent regulation and encodes a functional polypeptide": 20454–9। ডিওআই:10.1073/pnas.0708102104। পিএমআইডি 18042713। পিএমসি 2154452
 ।
।
- ↑ Dinger ME, Pang KC, Mercer TR, Mattick JS (নভেম্বর ২০০৮)। "Differentiating protein-coding and noncoding RNA: challenges and ambiguities": e1000176। ডিওআই:10.1371/journal.pcbi.1000176। পিএমআইডি 19043537। পিএমসি 2518207
 ।
।
- ↑ Saraiya AA, Wang CC (নভেম্বর ২০০৮)। "snoRNA, a novel precursor of microRNA in Giardia lamblia": e1000224। ডিওআই:10.1371/journal.ppat.1000224। পিএমআইডি 19043559। পিএমসি 2583053
 ।
।
- ↑ Ender C, Krek A, Friedländer MR, Beitzinger M, Weinmann L, Chen W, Pfeffer S, Rajewsky N, Meister G (নভেম্বর ২০০৮)। "A human snoRNA with microRNA-like functions": 519–28। ডিওআই:10.1016/j.molcel.2008.10.017। পিএমআইডি 19026782।
- ↑ Leygue E (আগস্ট ২০০৭)। "Steroid receptor RNA activator (SRA1): unusual bifaceted gene products with suspected relevance to breast cancer": e006। ডিওআই:10.1621/nrs.05006। পিএমআইডি 17710122। পিএমসি 1948073
 ।
।
- ↑ Zhang J, King ML (ডিসেম্বর ১৯৯৬)। "Xenopus VegT RNA is localized to the vegetal cortex during oogenesis and encodes a novel T-box transcription factor involved in mesodermal patterning": 4119–29। পিএমআইডি 9012531।
- ↑ Kloc M, Wilk K, Vargas D, Shirato Y, Bilinski S, Etkin LD (আগস্ট ২০০৫)। "Potential structural role of non-coding and coding RNAs in the organization of the cytoskeleton at the vegetal cortex of Xenopus oocytes": 3445–57। ডিওআই:10.1242/dev.01919
 । পিএমআইডি 16000384।
। পিএমআইডি 16000384।
- ↑ ক খ Jenny A, Hachet O, Závorszky P, Cyrklaff A, Weston MD, Johnston DS, Erdélyi M, Ephrussi A (আগস্ট ২০০৬)। "A translation-independent role of oskar RNA in early Drosophila oogenesis": 2827–33। ডিওআই:10.1242/dev.02456
 । পিএমআইডি 16835436।
। পিএমআইডি 16835436।
- ↑ Gultyaev AP, Roussis A (২০০৭)। "Identification of conserved secondary structures and expansion segments in enod40 RNAs reveals new enod40 homologues in plants": 3144–52। ডিওআই:10.1093/nar/gkm173। পিএমআইডি 17452360। পিএমসি 1888808
 ।
।
- ↑ Candeias MM, Malbert-Colas L, Powell DJ, Daskalogianni C, Maslon MM, Naski N, Bourougaa K, Calvo F, Fåhraeus R (সেপ্টেম্বর ২০০৮)। "P53 mRNA controls p53 activity by managing Mdm2 functions": 1098–105। ডিওআই:10.1038/ncb1770। পিএমআইডি 19160491।
- ↑ Gimpel M, Preis H, Barth E, Gramzow L, Brantl S (ডিসেম্বর ২০১২)। "SR1--a small RNA with two remarkably conserved functions": 11659–72। ডিওআই:10.1093/nar/gks895। পিএমআইডি 23034808। পিএমসি 3526287
 ।
।
- ↑ Francastel C, Hubé F (নভেম্বর ২০১১)। "Coding or non-coding: Need they be exclusive?": vi–vii। ডিওআই:10.1016/S0300-9084(11)00322-1। পিএমআইডি 21963143।
- ↑ Sempere LF, Sokol NS, Dubrovsky EB, Berger EM, Ambros V (জুলাই ২০০৩)। "Temporal regulation of microRNA expression in Drosophila melanogaster mediated by hormonal signals and broad-Complex gene activity": 9–18। ডিওআই:10.1016/S0012-1606(03)00208-2। পিএমআইডি 12812784।
- ↑ Adams BD, Furneaux H, White BA (মে ২০০৭)। "The micro-ribonucleic acid (miRNA) miR-206 targets the human estrogen receptor-alpha (ERalpha) and represses ERalpha messenger RNA and protein expression in breast cancer cell lines": 1132–47। ডিওআই:10.1210/me.2007-0022
 । পিএমআইডি 17312270।
। পিএমআইডি 17312270।
- ↑ Knoll M, Lodish HF, Sun L (মার্চ ২০১৫)। "Long non-coding RNAs as regulators of the endocrine system": 151–60। ডিওআই:10.1038/nrendo.2014.229। পিএমআইডি 25560704। পিএমসি 4376378
 ।
।
- ↑ Li W, Notani D, Ma Q, Tanasa B, Nunez E, Chen AY, Merkurjev D, Zhang J, Ohgi K, Song X, Oh S, Kim HS, Glass CK, Rosenfeld MG (জুন ২০১৩)। "Functional roles of enhancer RNAs for oestrogen-dependent transcriptional activation": 516–20। ডিওআই:10.1038/nature12210। পিএমআইডি 23728302। পিএমসি 3718886
 ।
।
- ↑ Pibouin L, Villaudy J, Ferbus D, Muleris M, Prospéri MT, Remvikos Y, Goubin G (ফেব্রুয়ারি ২০০২)। "Cloning of the mRNA of overexpression in colon carcinoma-1: a sequence overexpressed in a subset of colon carcinomas": 55–60। ডিওআই:10.1016/S0165-4608(01)00634-3। পিএমআইডি 11890990।
- ↑ Fu X, Ravindranath L, Tran N, Petrovics G, Srivastava S (মার্চ ২০০৬)। "Regulation of apoptosis by a prostate-specific and prostate cancer-associated noncoding gene, PCGEM1": 135–41। ডিওআই:10.1089/dna.2006.25.135। পিএমআইডি 16569192।
- ↑ ক খ Mourtada-Maarabouni M, Pickard MR, Hedge VL, Farzaneh F, Williams GT (জানুয়ারি ২০০৯)। "GAS5, a non-protein-coding RNA, controls apoptosis and is downregulated in breast cancer": 195–208। ডিওআই:10.1038/onc.2008.373
 । পিএমআইডি 18836484।
। পিএমআইডি 18836484।
- ↑ Dong XY, Guo P, Boyd J, Sun X, Li Q, Zhou W, Dong JT (আগস্ট ২০০৯)। "Implication of snoRNA U50 in human breast cancer": 447–54। ডিওআই:10.1016/S1673-8527(08)60134-4। পিএমআইডি 19683667। পিএমসি 2854654
 ।
।
- ↑ Christov CP, Trivier E, Krude T (মার্চ ২০০৮)। "Noncoding human Y RNAs are overexpressed in tumours and required for cell proliferation": 981–8। ডিওআই:10.1038/sj.bjc.6604254। পিএমআইডি 18283318। পিএমসি 2266855
 ।
।
- ↑ Farh KK, Grimson A, Jan C, Lewis BP, Johnston WK, Lim LP, Burge CB, Bartel DP (ডিসেম্বর ২০০৫)। "The widespread impact of mammalian MicroRNAs on mRNA repression and evolution": 1817–21। ডিওআই:10.1126/science.1121158। পিএমআইডি 16308420।
- ↑ Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, Bartel DP, Linsley PS, Johnson JM (ফেব্রুয়ারি ২০০৫)। "Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs": 769–73। ডিওআই:10.1038/nature03315। পিএমআইডি 15685193।
- ↑ Calin GA, Ferracin M, Cimmino A, Di Leva G, Shimizu M, Wojcik SE, Iorio MV, Visone R, Sever NI, Fabbri M, Iuliano R, Palumbo T, Pichiorri F, Roldo C, Garzon R, Sevignani C, Rassenti L, Alder H, Volinia S, Liu CG, Kipps TJ, Negrini M, Croce CM (অক্টোবর ২০০৫)। "A MicroRNA signature associated with prognosis and progression in chronic lymphocytic leukemia": 1793–801। ডিওআই:10.1056/NEJMoa050995। পিএমআইডি 16251535।
- ↑ Calin GA, Dumitru CD, Shimizu M, Bichi R, Zupo S, Noch E, Aldler H, Rattan S, Keating M, Rai K, Rassenti L, Kipps T, Negrini M, Bullrich F, Croce CM (নভেম্বর ২০০২)। "Frequent deletions and down-regulation of micro- RNA genes miR15 and miR16 at 13q14 in chronic lymphocytic leukemia": 15524–9। ডিওআই:10.1073/pnas.242606799। পিএমআইডি 12434020। পিএমসি 137750
 ।
।
- ↑ Hu Z, Chen J, Tian T, Zhou X, Gu H, Xu L, Zeng Y, Miao R, Jin G, Ma H, Chen Y, Shen H (জুলাই ২০০৮)। "Genetic variants of miRNA sequences and non-small cell lung cancer survival": 2600–8। ডিওআই:10.1172/JCI34934। পিএমআইডি 18521189। পিএমসি 2402113
 ।
।
- ↑ Shen J, Ambrosone CB, Zhao H (মার্চ ২০০৯)। "Novel genetic variants in microRNA genes and familial breast cancer": 1178–82। ডিওআই:10.1002/ijc.24008। পিএমআইডি 19048628।
- ↑ Sahoo T, del Gaudio D, German JR, Shinawi M, Peters SU, Person RE, Garnica A, Cheung SW, Beaudet AL (জুন ২০০৮)। "Prader-Willi phenotype caused by paternal deficiency for the HBII-85 C/D box small nucleolar RNA cluster": 719–21। ডিওআই:10.1038/ng.158। পিএমআইডি 18500341। পিএমসি 2705197
 ।
।
- ↑ Skryabin BV, Gubar LV, Seeger B, Pfeiffer J, Handel S, Robeck T, Karpova E, Rozhdestvensky TS, Brosius J (ডিসেম্বর ২০০৭)। "Deletion of the MBII-85 snoRNA gene cluster in mice results in postnatal growth retardation": e235। ডিওআই:10.1371/journal.pgen.0030235। পিএমআইডি 18166085। পিএমসি 2323313
 ।
।
- ↑ Ding F, Li HH, Zhang S, Solomon NM, Camper SA, Cohen P, Francke U (মার্চ ২০০৮)। "SnoRNA Snord116 (Pwcr1/MBII-85) deletion causes growth deficiency and hyperphagia in mice": e1709। ডিওআই:10.1371/journal.pone.0001709। পিএমআইডি 18320030। পিএমসি 2248623
 ।
।
- ↑ Ding F, Prints Y, Dhar MS, Johnson DK, Garnacho-Montero C, Nicholls RD, Francke U (জুন ২০০৫)। "Lack of Pwcr1/MBII-85 snoRNA is critical for neonatal lethality in Prader-Willi syndrome mouse models": 424–31। ডিওআই:10.1007/s00335-005-2460-2। পিএমআইডি 16075369।
- ↑ Bazeley PS, Shepelev V, Talebizadeh Z, Butler MG, Fedorova L, Filatov V, Fedorov A (জানুয়ারি ২০০৮)। "snoTARGET shows that human orphan snoRNA targets locate close to alternative splice junctions": 172–9। ডিওআই:10.1016/j.gene.2007.10.037। পিএমআইডি 18160232। পিএমসি 6800007
 ।
।
- ↑ Bolton PF, Veltman MW, Weisblatt E, Holmes JR, Thomas NS, Youings SA, Thompson RJ, Roberts SE, Dennis NR, Browne CE, Goodson S, Moore V, Brown J (সেপ্টেম্বর ২০০৪)। "Chromosome 15q11-13 abnormalities and other medical conditions in individuals with autism spectrum disorders": 131–7। ডিওআই:10.1097/00041444-200409000-00002। পিএমআইডি 15318025।
- ↑ Cook EH, Scherer SW (অক্টোবর ২০০৮)। "Copy-number variations associated with neuropsychiatric conditions": 919–23। ডিওআই:10.1038/nature07458। পিএমআইডি 18923514।
- ↑ Nakatani J, Tamada K, Hatanaka F, Ise S, Ohta H, Inoue K, Tomonaga S, Watanabe Y, Chung YJ, Banerjee R, Iwamoto K, Kato T, Okazawa M, Yamauchi K, Tanda K, Takao K, Miyakawa T, Bradley A, Takumi T (জুন ২০০৯)। "Abnormal behavior in a chromosome-engineered mouse model for human 15q11-13 duplication seen in autism": 1235–46। ডিওআই:10.1016/j.cell.2009.04.024। পিএমআইডি 19563756। পিএমসি 3710970
 ।
।
- ↑ Ziats MN, Rennert OM (মার্চ ২০১৩)। "Aberrant expression of long noncoding RNAs in autistic brain": 589–93। ডিওআই:10.1007/s12031-012-9880-8। পিএমআইডি 22949041। পিএমসি 3566384
 ।
।
- ↑ Ridanpää M, van Eenennaam H, Pelin K, Chadwick R, Johnson C, Yuan B, vanVenrooij W, Pruijn G, Salmela R, Rockas S, Mäkitie O, Kaitila I, de la Chapelle A (জানুয়ারি ২০০১)। "Mutations in the RNA component of RNase MRP cause a pleiotropic human disease, cartilage-hair hypoplasia": 195–203। ডিওআই:10.1016/S0092-8674(01)00205-7। পিএমআইডি 11207361।
- ↑ Martin AN, Li Y (মার্চ ২০০৭)। "RNase MRP RNA and human genetic diseases": 219–26। ডিওআই:10.1038/sj.cr.7310120
 । পিএমআইডি 17189938।
। পিএমআইডি 17189938।
- ↑ Kavadas FD, Giliani S, Gu Y, Mazzolari E, Bates A, Pegoiani E, Roifman CM, Notarangelo LD (ডিসেম্বর ২০০৮)। "Variability of clinical and laboratory features among patients with ribonuclease mitochondrial RNA processing endoribonuclease gene mutations": 1178–84। ডিওআই:10.1016/j.jaci.2008.07.036। পিএমআইডি 18804272।
- ↑ Faghihi MA, Modarresi F, Khalil AM, Wood DE, Sahagan BG, Morgan TE, Finch CE, St Laurent G, Kenny PJ, Wahlestedt C (জুলাই ২০০৮)। "Expression of a noncoding RNA is elevated in Alzheimer's disease and drives rapid feed-forward regulation of beta-secretase": 723–30। ডিওআই:10.1038/nm1784। পিএমআইডি 18587408। পিএমসি 2826895
 ।
।
- ↑ Mencía A, Modamio-Høybjør S, Redshaw N, Morín M, Mayo-Merino F, Olavarrieta L, Aguirre LA, del Castillo I, Steel KP, Dalmay T, Moreno F, Moreno-Pelayo MA (মে ২০০৯)। "Mutations in the seed region of human miR-96 are responsible for nonsyndromic progressive hearing loss": 609–13। ডিওআই:10.1038/ng.355। পিএমআইডি 19363479।
- ↑ Lewis MA, Quint E, Glazier AM, Fuchs H, De Angelis MH, Langford C, van Dongen S, Abreu-Goodger C, Piipari M, Redshaw N, Dalmay T, Moreno-Pelayo MA, Enright AJ, Steel KP (মে ২০০৯)। "An ENU-induced mutation of miR-96 associated with progressive hearing loss in mice": 614–8। ডিওআই:10.1038/ng.369। পিএমআইডি 19363478। পিএমসি 2705913
 ।
।
- ↑ Soukup GA (জুন ২০০৯)। "Little but loud: small RNAs have a resounding affect on ear development": 104–14। ডিওআই:10.1016/j.brainres.2009.02.027। পিএমআইডি 19245798। পিএমসি 2700218
 ।
।
- ↑ Carter RJ, Dubchak I, Holbrook SR (অক্টোবর ২০০১)। "A computational approach to identify genes for functional RNAs in genomic sequences": 3928–38। ডিওআই:10.1093/nar/29.19.3928। পিএমআইডি 11574674। পিএমসি 60242
 ।
।
- ↑ Pedersen JS, Bejerano G, Siepel A, Rosenbloom K, Lindblad-Toh K, Lander ES, Kent J, Miller W, Haussler D (এপ্রিল ২০০৬)। "Identification and classification of conserved RNA secondary structures in the human genome": e33। ডিওআই:10.1371/journal.pcbi.0020033। পিএমআইডি 16628248। পিএমসি 1440920
 ।
।
- ↑ Thomas JM, Horspool D, Brown G, Tcherepanov V, Upton C (জানুয়ারি ২০০৭)। "GraphDNA: a Java program for graphical display of DNA composition analyses": 21। ডিওআই:10.1186/1471-2105-8-21। পিএমআইডি 17244370। পিএমসি 1783863
 ।
।
- ↑ Brosius J, Raabe CA (ফেব্রুয়ারি ২০১৫)। "What is an RNA? A top layer for RNA classification": 140–4। ডিওআই:10.1080/15476286.2015.1128064। পিএমআইডি 26818079। পিএমসি 4829331
 ।
।
- ↑ Ji, Zhe; Song, Ruisheng (২০১৫-১২-১৯)। "Many lncRNAs, 5'UTRs, and pseudogenes are translated and some are likely to express functional proteins" (ইংরেজি ভাষায়): e08890। আইএসএসএন 2050-084X। ডিওআই:10.7554/eLife.08890। পিএমআইডি 26687005। পিএমসি 4739776
 ।
।
- ↑ Tosar, Juan Pablo; Rovira, Carlos (২০১৮-০১-২২)। "Non-coding RNA fragments account for the majority of annotated piRNAs expressed in somatic non-gonadal tissues" (ইংরেজি ভাষায়): 2। আইএসএসএন 2399-3642। ডিওআই:10.1038/s42003-017-0001-7। পিএমআইডি 30271890। পিএমসি 6052916
 ।
।
- ↑ Housman, Gali; Ulitsky, Igor (জানুয়ারি ২০১৬)। "Methods for distinguishing between protein-coding and long noncoding RNAs and the elusive biological purpose of translation of long noncoding RNAs": 31–40। আইএসএসএন 0006-3002। ডিওআই:10.1016/j.bbagrm.2015.07.017। পিএমআইডি 26265145।
<references>-এ সংজ্ঞায়িত "pmid17571346" নামসহ <ref> ট্যাগ পূর্ববর্তী লেখায় ব্যবহৃত হয়নি।বহিঃসংযোগ[সম্পাদনা]
( ওয়েব্যাক মেশিন অনুলিপি)
- আরফাম ডেটাবেস - সম্পর্কিত এনসিআরএনএ-র শত শত পরিবারের একটি সংক্ষিপ্ত তালিকা
- NONCODE.org - সমস্ত ধরনের ননকোডিং আরএনএর একটি বিনামূল্যে ডাটাবেস (টিআরএনএ এবং আরআরএনএ বাদে)
- আরএনএকন এনসিআরএনএ বিএমসি জিনোমিক্স 2014, 15: 127 এর পূর্বাভাস এবং শ্রেণিবিন্যাস
- এনকোড থ্রেড এক্সপ্লোরার নন-কোডিং আরএনএ বৈশিষ্ট্য। প্রকৃতি (জার্নাল)
- নন-কোডিং আরএনএ ডেটাবেসেস রিসোর্স (এনআরডিআর) - ইন্টারনেটে উপলব্ধ নন-কোডিং আরএনএ ডাটাবেসগুলির সাথে সম্পর্কিত ডেটাগুলির একটি সঞ্চিত উৎস
- DASHR - ছোটো নন-কোডিং আরএনএ বায়োইনফরম্যাটিক্স 2018 এর একটি ডাটাবেস
